T细胞受体(TCR)在适应性免疫系统中扮演着核心角色,能够识别并响应病原体及异常细胞。TCR的多样性源自V(D)J重排及α/β链配对,尤其是识别抗原的互补决定区3(CDR3),使TCR能够识别广泛的抗原。因此,表征抗原受体库并理解其在疾病进展中的动态变化对疫苗设计和精准免疫治疗至关重要。近年来,已经开发了多种从单细胞RNA测序(scRNA-seq)数据集中构建TCR的方法,每种方法具有其不同的特征和功能。然而,在不同条件下对这些方法性能的全面评估仍然缺乏。
近日,浙江大学爱丁堡大学联合学院(ZJE)刘琬璐研究员团队,在Genomics, Proteomics & Bioinformatics (GPB)期刊发表了题为:Evaluation of T Cell Receptor Construction Methods from scRNA-Seq Data的研究论文。该研究工作采用多种类型的数据集对七种TCR重构方法进行了全面性能评估,此外,研究团队还开发了YASIM-scTCR模拟工具,可根据用户设置的参数(如测序深度和读长)生成包含TCR和非TCR序列的scTCR-seq数据,从而实现对不同方法在准确性和灵敏度上的精确对比分析。
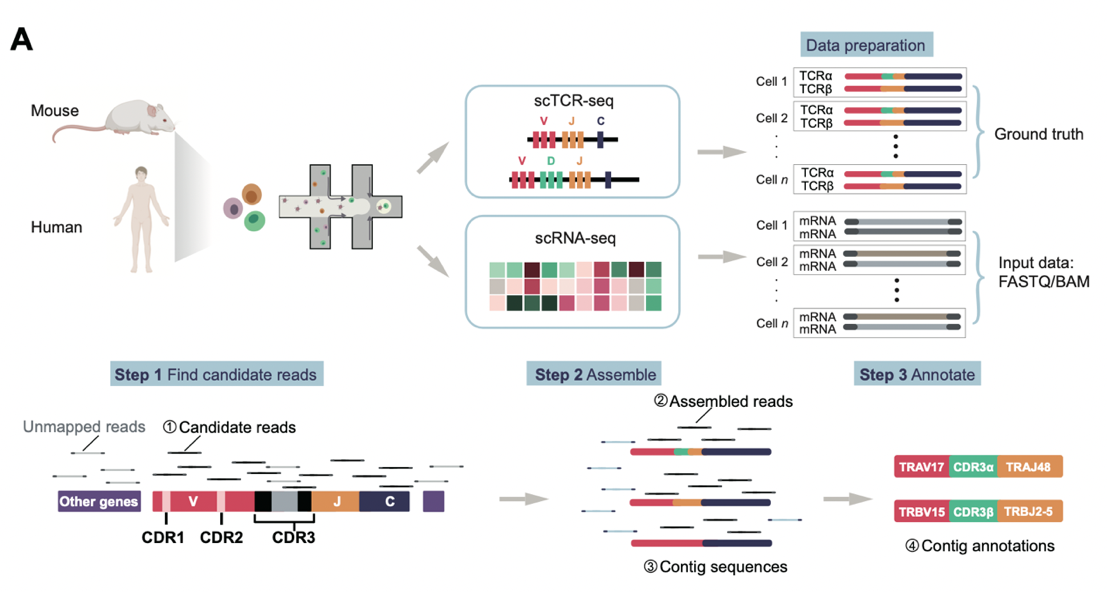
该研究评估了七种TCR重建方法的性能,使用了包含人类和小鼠的scRNA-seq和scTCR-seq数据集。通过对比10X scRNA-seq数据和配对的scTCR-seq数据,评估了各方法的准确性和灵敏度。TCR重建方法通常分为三个步骤:1.从FASTQ或BAM文件中寻找候选TCR序列。 2.使用算法组装TCR序列。 3.识别V(D)J片段并注释CDR3区。
为了能够更深入和全面的探索影响从scRNA-seq中重组TCR结果的原因,该研究使用YASIM-scTCR模拟工具生成了包含TCR衍生读取的scTCR-seq模拟数据,并对各方法在模拟数据上的表现进行了评估。结果显示,TRUST4、TraCeR和ImRep在候选读取识别方面表现优异,而DeRR在TCR组装的准确性上相对较弱。此外,各方法在模拟数据上的表现远远优于真实数据,这意味着影响从真实 scRNA-seq 数据中重组 TCR 的主要原因在于 scRNA-seq 所含的 TCR 读取不充分,而非软件自身算法的局限性。
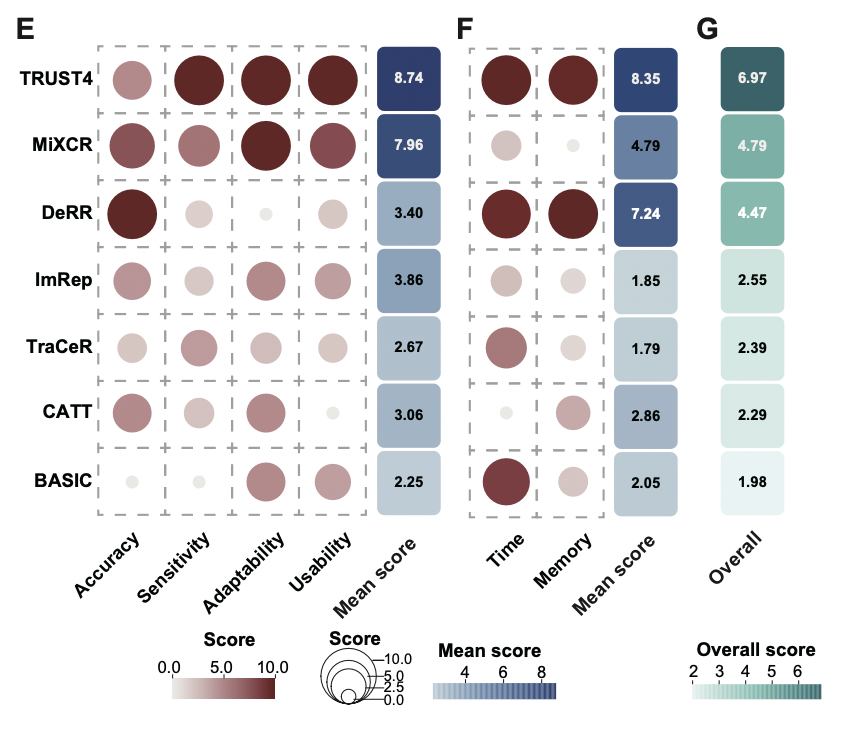
于具备不同测序深度与长度的模拟数据对比过程中,能够发现不同的测序深度会对 TCR 组装性能产生极为显著的影响。最后,综合评估包括准确性、灵敏度、适应性、易用性、时间和内存消耗等六个方面,最终TRUST4排名最高,其次是MiXCR和DeRR。
作者及资助信息:
浙江大学爱丁堡大学联合学院刘琬璐研究员为本文通讯作者;浙江大学丁堡大学联合学院在读博士生田若楠、爱丁堡大学硕士俞哲健为本文共同第一作者。本研究得到了腾讯AI Lab首席科学家姚建华、研究员何冰与赵宇,以及浙江大学医学院鲁林荣教授的共同参与,同时ZJE 2019级生物信息学专业本科生蔡铄也积极参与了课题的研究工作。该项目得到了国家自然科学基金和腾讯AI Lab犀牛鸟专项研究计划的资助支持。







