近日,浙江大学爱丁堡大学联合学院(ZJE)刘琬璐团队与浙江大学医学院鲁林荣团队,联合腾讯生命科学实验室等多家单位,在国际学术期Cell Discovery发表题为A pan-disease and population-level single-cell TCRαβ repertoire reference的研究论文。该研究首次构建了一个覆盖跨人群、多疾病的单细胞T细胞受体(TCRαβ)参考图谱,并开发了创新计算框架 TCR-DeepInsight,为系统解析T细胞受体与疾病的关系提供了全新的工具和资源。

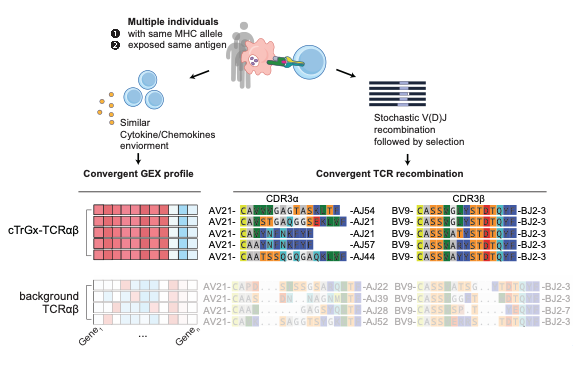
T细胞是机体适应性免疫系统的核心组成部分,其表面的T细胞受体(TCR)通过识别抗原肽-MHC复合物介导病原体清除和肿瘤免疫监视。由于TCR的α、β链在发育过程中经历高度随机的V(D)J重排,形成了极其庞大的序列多样性。然而,在不同个体之间,仍存在部分序列相同或高度相似的“公共TCR” (Public TCR),它们往往与常见病毒感染或特定免疫反应密切相关。
尽管近年来单细胞测序技术的快速发展使得同时获取TCR序列和转录组信息成为可能,但如何在更大的人群和疾病范围内整合分析这些信息,系统揭示TCR多样性与免疫表型的关联,仍是免疫学领域的关键挑战。
研究团队整合了来自70项研究、1017个生物样本、583名个体及46种疾病的单细胞TCR测序数据,共包含2,298,876个高置信度T细胞,并为每个细胞提供了全长TCRαβ序列、转录组表达谱及HLA基因型信息。
该数据现已纳入团队前期建立的人类抗原受体数据库huARdb 2.0(Wu et al., 2022, Nucleic Acids Research),为全球研究者提供可检索和比对的单细胞TCRαβ资源。
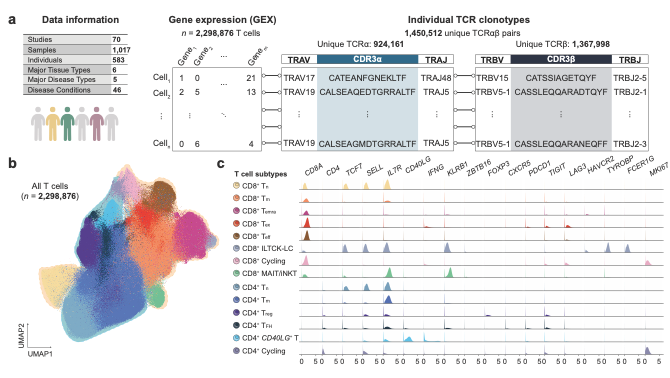
通过系统分析,研究发现TCR序列特征与CD4⁺、CD8⁺谱系分化密切相关,特定V/J基因的组合及CDR区域的氨基酸组成在不同谱系中存在显著偏好。这一结果揭示了TCR与MHC限制性识别之间的遗传学基础。
研究在全人群范围内鉴定出近3000个公共TCRαβ克隆型,它们往往与高克隆扩增度及共享的HLA等位基因相关,且可能识别常见病毒(如EB病毒、巨细胞病毒、流感病毒)来源的抗原肽,反映出人群层面免疫应答的共性规律。
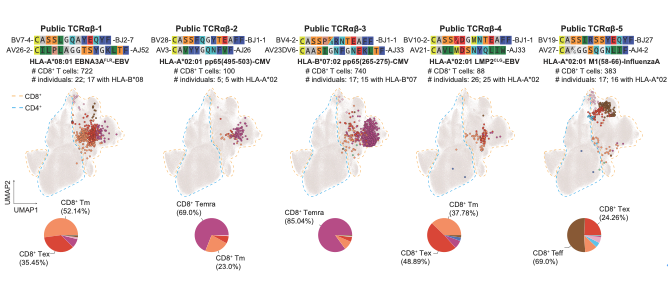
团队开发的TCR-DeepInsight算法利用深度学习模型(BERT与变分自编码器(Variational autoencoder, VAE)),在百万级单细胞数据上实现TCR序列与转录组信息的联合表示,能够识别具有相似功能特征的TCR簇群,并自动发现HLA共享型或疾病相关型TCR。该框架支持研究者将自身数据快速映射到参考图谱中,实现跨疾病、跨人群的免疫比较分析。
该研究的发布,标志着单细胞免疫组学进入了人群级整合与智能分析的新阶段。
通过建立覆盖多疾病与多个体的TCRαβ全景参考图谱,研究团队为理解T细胞多样性、免疫记忆形成及疾病相关免疫反应提供了关键基础,也为未来TCR-T细胞治疗、疫苗设计及免疫疾病诊断提供了重要的数据支撑。
未来,研究团队计划进一步扩充参考数据库的样本数量与疾病类型,并结合人工智能预测模型,推动精准免疫学和个体化治疗的发展。
ZJE刘琬璐研究员,浙江大学医学院鲁林荣教授为本文通讯作者;ZJE院双博士项目在读博士生薛子为,浙江大学医学院博士研究生吴利则,ZJE毕业生高兵博士为本文共同第一作者。本研究得到了腾讯生命科学实验室的共同参与,同时ZJE 2022级生物信息学专业本科生陈奕儒、祁意城和董天泽也参与了课题的研究工作。该项目得到了“前沿生物技术”国家重点研发专项青年科学家项目等基金的大力支持,为研究的顺利开展提供了重要保障。
课题组介绍
About lab
刘琬璐团队主要通过单细胞空间多组学以及深度学习算法开发,聚焦生物信息学以及T细胞计算免疫学研究。已以通讯作者(含共同)在Cell、Nature Methods, Nature Communications、Nucleic Acids Research等发表研究论文50篇,申请国家发明专利4项(获授权1项),主持国家自然科学基金面上项目、腾讯AI Lab犀牛鸟专项研究计划,入选浙江省和国家级青年人才计划,担任中国生物化学与分子生物学会分子免疫学专业分会委员、中国细胞生物学学会免疫细胞生物学分会委员、浙江省生物信息学学会理事会理事、浙江省生物信息学学会青年专业委员会委员、英国爱丁堡大学Honorary lecturer等学术任职。
邮箱:wanluliu@intl.zju.edu.cn;
实验室网址:https://labw.org







